RNA-Seq 分析流程
RNA-Seq是科研和生产领域常用的分析流程。其分析相比DNA分析而言,流程相对复杂。用户如果Linux命令行执行,分析任务需要投入大量精力,对作业调度和作业监控进行设计,从而加大了开发难度。
RNA-Seq 数据分析概述
由于RNA-Seq 分析在一次实验中可以对样本的基因转录进行检测。因此其应用领域非常广泛,而针对这种情况,在分析工具上也存在多种选择。例如,在癌症研究领域,STAR 是较为常见的比对工具,主要用于融合基因检测,而在更多的场景中hisat2 由于其比对过程中结合SNP 等信息,也是应用广泛的比对工具。
针对这种情况,WDL 的灵活语法,提供了非常好的功能模块化管理功能,用户可以将不同的功能进行模块化处理,从而可以轻松实现代码地替换和重复利用。
Achelous 平台上的RNA-Seq 分析
针对流程复杂、监控困难的问题,在Achelous平台上使用WDL脚本进行流程编排,是用户最佳的解决方案。极道提供了基于Achelous平台的RNA-Seq分析示例流程,用户可以通过资料下载页面进行下载,其内容涵盖以下功能:
- 数据指控、基因组比对、转录本拼接等基础分析
- 基因表达量分析以及差异基因筛选
- 转录组可变剪切分析
- 样本SNP分析
- 差异基因功能注释分析
Achelous 流程可视化界面
对于RNA-Seq这类相对复杂的流程,可以通过Achelous 中的Biostack 前端界面进行流程的拓扑结构展示,并且在任务提交过程中。会动态显示每个任务的运行状态,使用户可以轻松了解各任务的运行情况。
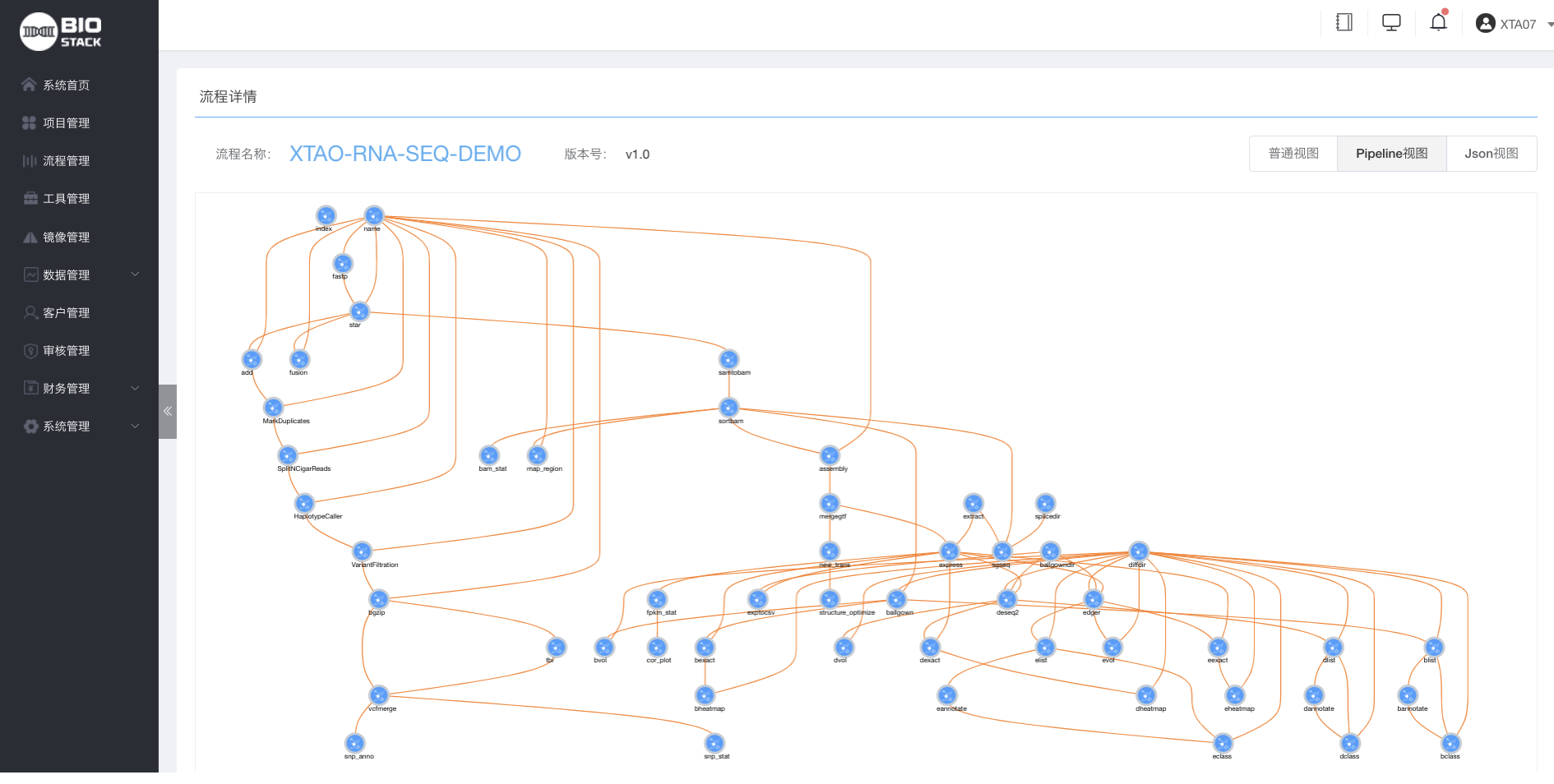
用户可以基于该流程,针对性的对其中各分析模块中使用的工具进行替换。
